Projekt Biochemie
PD Dr. Helmut Pospiech hat das Forschungsgebiet Biochemie am FLI und der FSU bis zur Neubesetzung der Professur Biochemie des Alterns durch Frau Prof. Janine Kirstein in 2023 weitergeführt. Er ist jetzt am .
Im Fokus der Projektgruppe Biochemie steht die Untersuchung von DNA-Replikation und Erhalt der genomischen Integrität im Alter und bei alternsbezogenen Krankheiten. Wir untersuchen die Mechanismen von DNA-Replikation, -Reparatur und -Schadensantwort, sowie die Folgen von Störungen in diesen Abläufen.
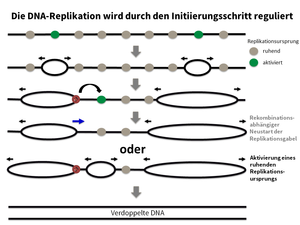
Die DNA-Replikation, d.h. die Verdopplung von genetischer Information, ist ein grundlegender Prozess aller lebenden Zellen. Eine genaue und vollständige DNA-Replikation ist daher die Voraussetzung für die erfolgreiche Teilung und das Wachstum von Zellen. Deshalb ist es wenig überraschend, dass dieser Vorgang mit erstaunlicher Plastizität und Genauigkeit vonstattengeht. Die DNA-Replikation wird von Prozessen wie der DNA-Reparatur und der DNA-Schadensantwort unterstützt, um auftretende Schäden optimal zu reparieren. Trotzdem kommen Fehler in der DNA-Replikation vor, die sich über die Zeit hinweg anhäufen. Diese führen in Zellen und Geweben zu Störungen, die dem Alternsprozess und der Krebsentwicklung zu Grunde liegen. Dies ist vor allem für besonders große und langlebige Organismen, wie z.B. den Menschen, problematisch.
Wir beschäftigen uns mit vielfältigen Aspekten der DNA-Replikation, -Reparatur und -Schadensantwort, genauer mit folgenden Themen:

AgingComb - Untersuchung der Genomalterung mittels Hochdurchsatzanalysen von DNA-Replikation und Rekombination
Zahlreiche Studien der letzten Jahre weisen auf die Bedeutung der fortschreitenden Schädigung des Genoms bei Alterungsprozessen hin. Allerdings sind die molekularen Mechanismen, die im Alter die Genomintegrität verletzen, nur teilweise verstanden. Im Leibniz ScienceCampus Regenerative Aging kooperieren dazu die Arbeitsgruppen von Holger Bierhoff (Friedrich-Schiller-Universität Jena), Thomas Liehr, Florian Heidel (beide Universitätsklinikum Jena) und Helmut Pospiech (Leibniz-Institut für Alternsforschung).
Das kooperative Projekt AgingComb beleuchtet zwei zentrale Prozesse des DNA-Metabolismus, die sich auf die Integrität des Genoms auswirken: die Replikation und die Rekombination. Mittels der innovativen ‚Molecular Combing‘-Technik können Veränderungen in der Genomstruktur im Hochdurchsatzverfahren dokumentiert werden. Hier kommt die Fiber Combing Plattform der Firma Genomic Vision zum Einsatz. Die dabei generierten Datensätze erlauben neue Einblicke in die Stabilität des Genoms und die molekularen Mechanismen, die die DNA-Replikation und -Rekombination im Zuge der Alterung beeinträchtigen. Über die Bündelung unserer Expertisen aus Grundlagenforschung und Klinischer Praxis wollen wir weiterhin den Nutzen für klinische Anwendungen unterstützen.
Das Projekt AgingComb wird aus Mitteln des Europäischen Fonds für Regionale Entwicklung (EFRE) der Europäischen Union finanziert, die vom Thüringer Ministerium für Wirtschaft, Wissenschaft und Digitale Gesellschaft bereitgestellt werden.
Kontakt

Helmut Pospiech
Ehemaliger Projektleiter
+49 3641 65-6290
helmut.pospiech@~@leibniz-fli.de
Wie führen erbliche Mutationen in bestimmten Genen (dem „BRCA-Stoffwechselweg“) zu einem frühen Auftreten von Brustkrebs? Inwiefern führt ein Ungleichgewicht zwischen DNA-Replikation und -Rekombination zu genomischer Instabilität?
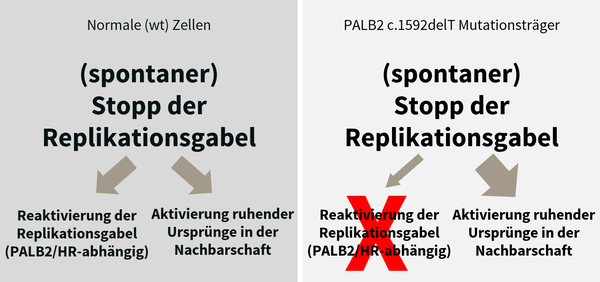
Brustkrebs ist der am häufigsten auftretende Krebs bei Frauen in Industrieländern. Heterozygote Mutationen in bestimmten Genen erhöhen das Brustkrebsrisiko und setzen das Alter für das Auftreten von Brustkrebs herab. Die meisten der bisher bekannten Brustkrebsgene spielen eine Rolle im sogenannten BRCA-Stoffwechselweg, der die homologe Rekombination nach DNA-Doppelstrangbrüchen und während der DNA-Replikation reguliert. Wir konnten bei finnischen Trägern einer PALB2-Mutation nachweisen, dass bereits der Verlust einer der beiden Kopien dieser Gene zu einem DNA-Replikationsstress-Phänotyp sowie zu Defekten bei der Aufrechterhaltung von Zellzyklus-Kontrollpunkten führt. Diese führen zusammen zu genomischer Instabilität; ein früher Vorantreiber von Krebs (Nikkilä et al., 2013; Obermeier et al., 2016). In Zusammenarbeit mit deutschen und internationalen Partnern untersuchen wir die funktionellen Konsequenzen weiterer erblicher Brustkrebsmutationen. In einem DFG-geförderten Projekt untersuchen wir genauer, welche Auswirkungen eine Reduktion von Faktoren, die am BRCA-Stoffwechselweg beteiligt sind, auf die DNA-Replikation, homologe Rekombination, DNA-Schadensantwort und genomische Stabilität hat.
Quelle:
Nikkilä, J. et al. (2013). Heterozygous mutations in PALB2 cause DNA replication and damage response defects, Nat Commun, 4, 2578. [PubMed]
Obermeier, K. et al. (2016). Heterozygous PALB2 c.1592delT mutation channels DNA double-strand break repair into error-prone pathways in breast cancer patients, Oncogene, 35(29), 3796-806. [PubMed]
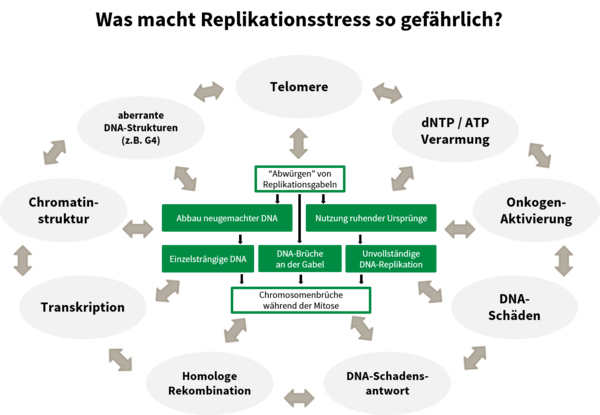
Durch welche Mechanismen verursacht DNA-Replikationsstress eine Genomdestabilisierung?
Der DNA-Replikationsstress wurde als Schlüsselfaktor für genomische Instabilität und Krebs identifiziert. Die Anzahl von DNA-Replikationsrunden von Stammzellen im Körper und damit das Alter, sind ein wesentlicher Einflussfaktor für das Krebsrisiko. Die Ursachen für DNA-Replikationsstress sind vielfältig, ebenso wie die damit verbundenen Phänotypen. Wir haben verschiedene Fälle von DNA-Replikationsstress untersucht, ausgelöst zum Beispiel durch Störungen der DNA-Replikation (Köhler et al., 2016; Sokka et al., 2018), der homologen Rekombination (Nikkilä et al., 2013; Parplys et al., 2015), der DNA-Schadenstoleranz (Hampp et al., 2016) oder metabolische Defekte (Pai et al., 2016), um unsere Erkenntnisse zu den Ursachen, Phänotypen und den daraus resultierenden Folgen eines DNA-Replikationsstress zu erweitern.
Quelle:
Hampp, S. et al. (2016). A DNA damage tolerance pathway involving DNA polymerase iota and the tumor suppressor p53 regulates DNA replication fork progression, Proc Natl Acad Sci U S A, 113(30), E4311-9. [PubMed]
Köhler, C. et al. (2016). Cdc45 is limiting for replication initiation in humans, Cell Cycle, 15(7), 974-85. [PubMed]
Nikkilä, J. et al. (2013). Heterozygous mutations in PALB2 cause DNA replication and damage response defects, Nat Commun, 4, 2578. [PubMed]
Pai, G. M. et al. (2016). TSC loss distorts DNA replication programme and sensitises cells to genotoxic stress, Oncotarget, 7(51), 85365-80. [PubMed]
Parplys, A. C. et al. (2015). High levels of RAD51 perturb DNA replication elongation and cause unscheduled origin firing due to impaired CHK1 activation, Cell Cycle, 14(19), 3190-202. [PubMed]
Sokka, M. et al. (2018). The ATR-activation domain of TopBP1 is required for the suppression of origin firing during the S phase, Int J Mol Sci, 19(8), pii: E2376. [PubMed]
Welche Obergrenze haben Mutationsraten in menschlichen Zellen und wie können Mutationsraten im Alter und bei Krebs kontrolliert werden?
Aktuell beschäftigen wir uns mit den Mutationsraten, die während der DNA-Replikation in menschlichen Zellen auftreten. Dabei versuchen wir die obere Grenze an Mutationen zu definieren, die von menschlichen Zellen toleriert werden können, und die spezifischen Merkmale von (Krebs-)Zellen, die mit extrem hohen Mutationsraten existieren, zu charakterisieren. Wir erforschen außerdem, wie sich Mutationen im Alter ansammeln und wie sich bei Organismen mit unterschiedlicher Lebenserwartung die Mutationsraten unterscheiden. Darüber hinaus suchen wir neue Wege, mit denen die Mutationsraten positiv und bei der Behandlung von Krebs negativ beeinflusst werden können. Außerdem versuchen wir nach Wegen, die Mutationsakkumulation beim Altern zu verringern.
Welche molekularen Funktionen haben individuelle Replikationsproteine?
Ein langjähriges Forschungsinteresse der Projektgruppe liegt bei der Charakterisierung von Faktoren, die bei DNA-Replikation und -Rekombination eine Rolle spielen. Diese sind zum Beispiel die replikativen DNA-Polymerasen α, δ und ε (Vaara et al., 2012; Villani et al., 2014; Itkonen et al., 2016), der Helikase Co-Faktor CDC45 (Szambowska et al., 2014, 2017; Köhler et al., 2016) und die DNA-Helikasen RecQL4 (Ohlenschläger et al., 2012; Keller et al., 2014) und S100A11 (Foertsch et al., 2016). Vor kurzem haben wir einen neuen potenziellen Replikationsfaktor identifiziert, den wir PN70 getauft haben. Dieser wird derzeit detailliert funktionell charakterisiert.
Quelle:
Foertsch, F. et al. (2016). S100A11 plays a role in homologous recombination and genome maintenance by influencing the persistence of RAD51 in DNA repair foci, Cell Cycle, 15(20), 2766-79. [PubMed]
Itkonen, H. M. et al. (2016). Human DNA polymerase α interacts with mismatch repair proteins MSH2 and MSH6, FEBS Lett, 590(23), 4233-41. [PubMed]
Keller, H. et al. (2014). The intrinsically disordered amino-terminal region of human RecQL4 : multiple DNA-binding domains confer annealing , strand exchange and G4 DNA binding, Nucleic Acids Res, 42(20), 12614-27. [PubMed]
Köhler, C. et al. (2016). Cdc45 is limiting for replication initiation in humans, Cell Cycle, 15(7), 974-85. [PubMed]
Ohlenschläger, O. et al. (2012). The N-terminus of the human RecQL4 helicase is a homeodomain-like DNA interaction motif, Nucleic Acids Res, 40(17), 8309-24. [PubMed]
Szambowska, A. et al. (2014). DNA binding properties of human Cdc45 suggest a function as molecular wedge for DNA unwinding, Nucleic Acids Res, 42(4), 2308-19. [PubMed]
Szambowska, A. et al. (2017). Cdc45-induced loading of human RPA onto single-stranded DNA, Nucleic Acids Res, 45(6), 3217-30. [PubMed]
Vaara, M. et al. (2012). Segregation of replicative DNA polymerases during S phase: DNA polymerase ε, but not DNA polymerases α/δ, are associated with lamins throughout S phase in human cells, J Biol Chem, 287(40), 33327-38. [PubMed]
Villani, G. et al. (2014). Gap-directed translesion DNA synthesis of an abasic site on circular DNA templates by a human replication complex, PLoS One, 9(4), e93908. [PubMed]
Team*
| Name | Telefon | Tätigkeit |
|---|
* unvollständig aufgrund DSGVO